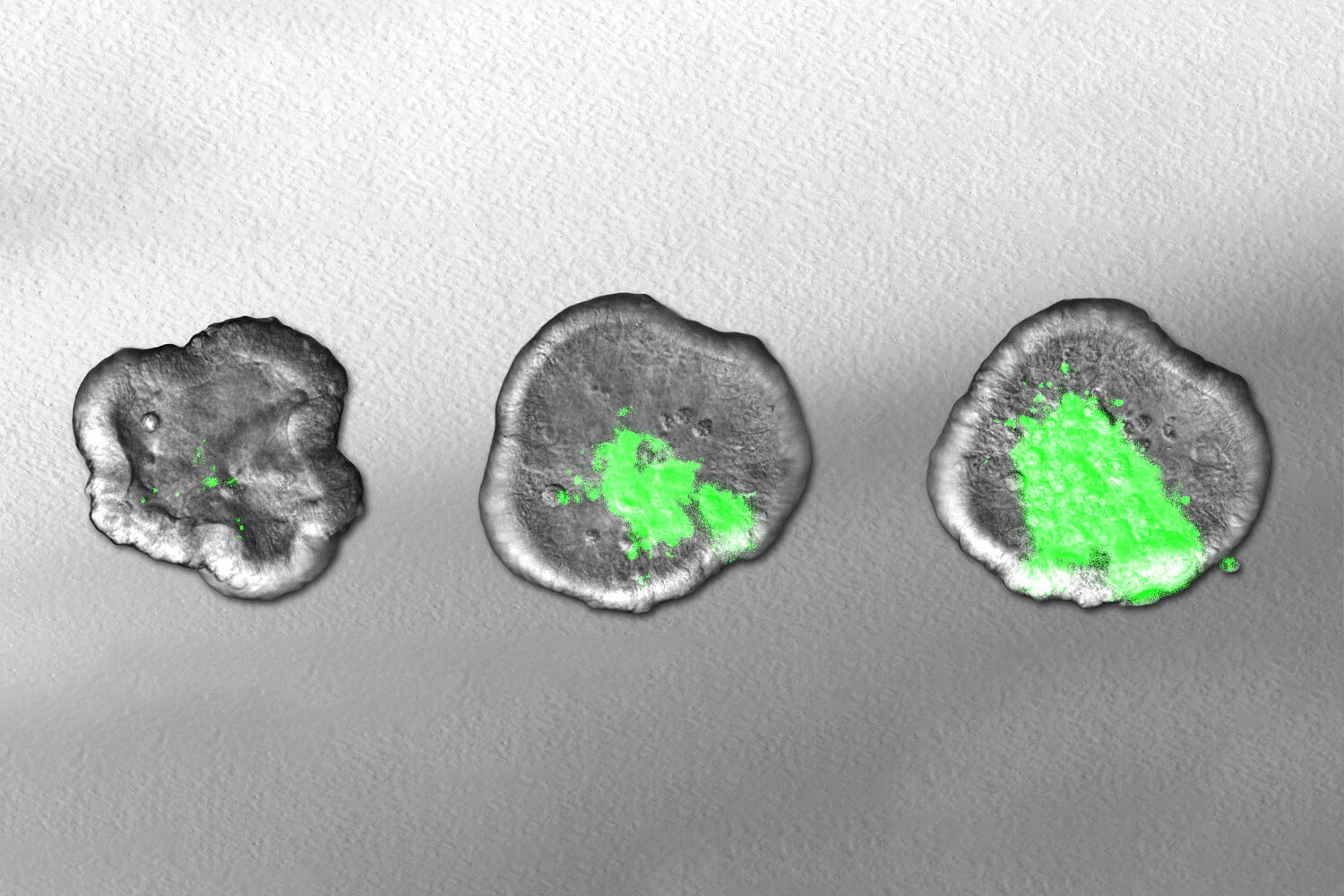
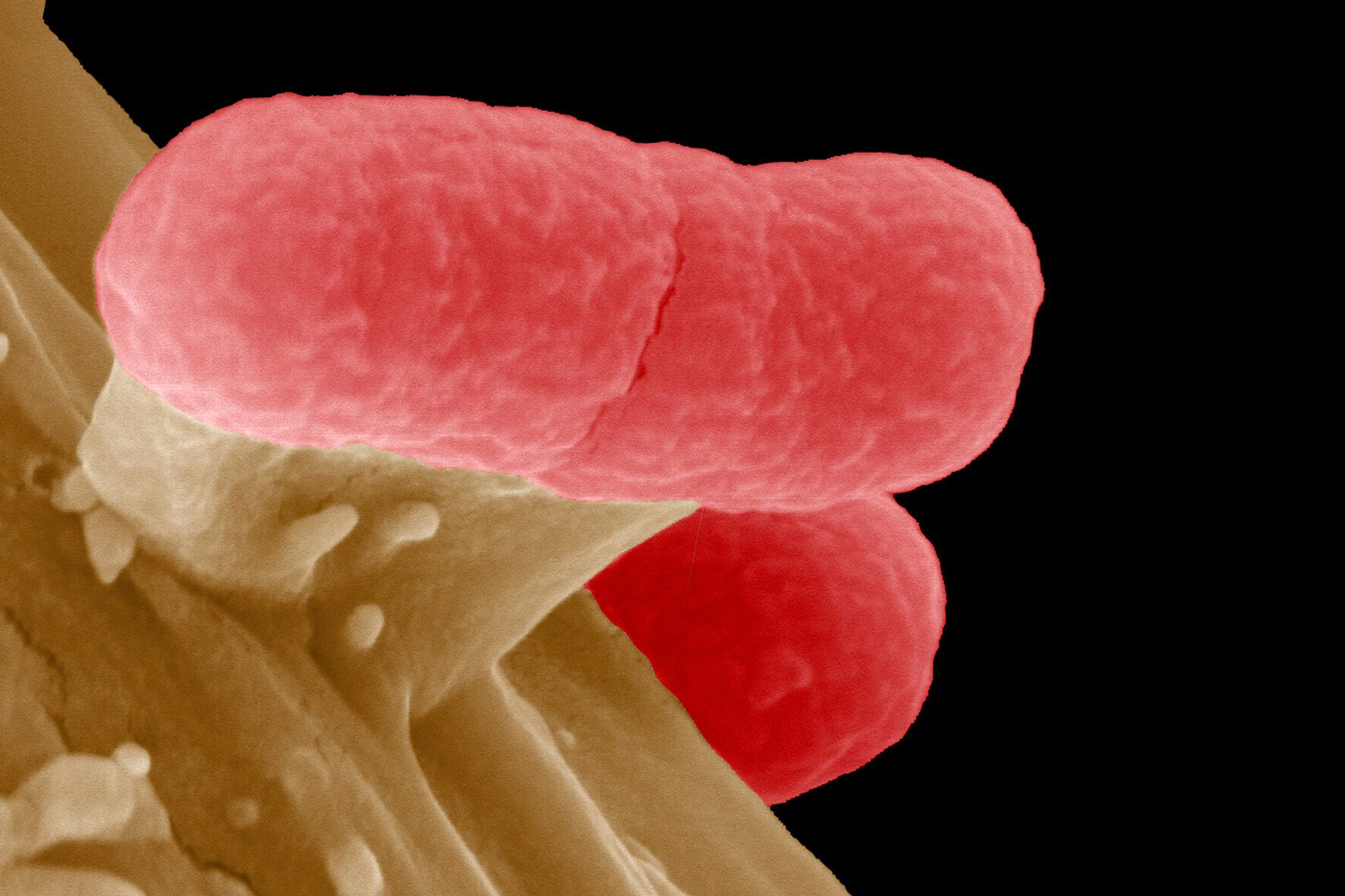
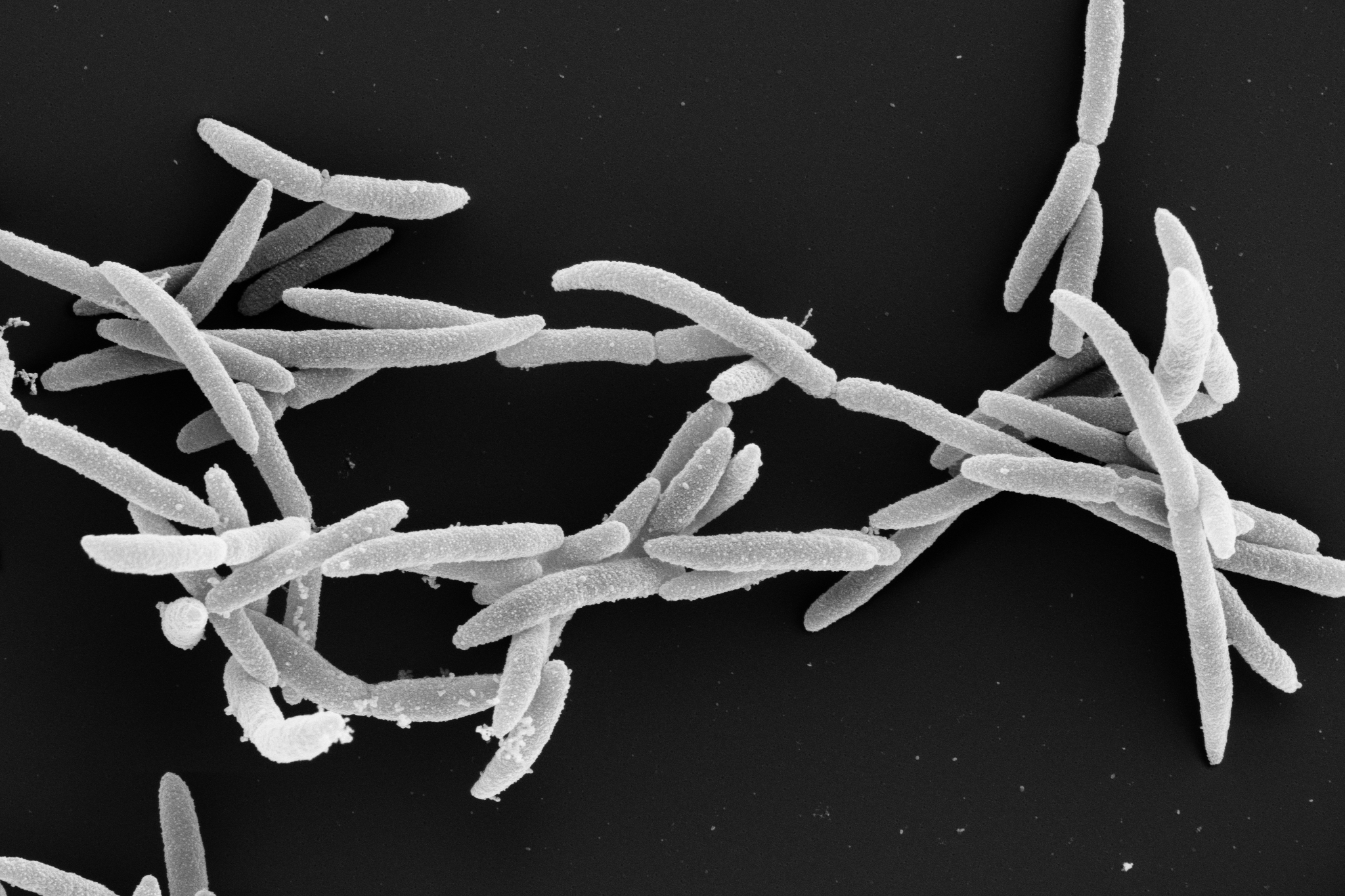
Zu verstehen, wie menschenspezifische Bakterien uns genau krankmachen, ist eine Herausforderung. In einer neuen Studie, die heute in Nature Genetics veröffentlicht wurde, zeigen Forschende, dass sich anhand von gezüchteten Mini-Organen, sogenannten Organoiden, nachvollziehen lässt, wie Mikroben die Darmschleimhaut besiedeln. Das Team des Helmholtz-Instituts für RNA-basierte Infektionsforschung (HIRI) in Würzburg – einem Standort des Braunschweiger Helmholtz-Zentrums für Infektionsforschung (HZI) in Kooperation mit der Julius-Maximilians-Universität Würzburg (JMU) – und der Universität Uppsala konzentrierte sich dabei insbesondere auf Shigella flexneri. Dieses Bakterium verursacht beim Menschen schwere Darmentzündungen und ist jährlich für zahlreiche Todesfälle verantwortlich, insbesondere bei Kleinkindern.
„Unsere Ergebnisse liefern ein realistischeres Bild der Faktoren, die die Besiedlung unseres Darms mit Shigella beeinflussen. Künftig könnten sie neue medizinische Behandlungsansätze ermöglichen“, sagt Lars Barquist, assoziierter Wissenschaftler am HIRI, Professor an der Universität Toronto in Kanada und Ko-Seniorautor der Studie. „Zum ersten Mal konnten wir die Gene kartieren, die Shigella für eine Infektion braucht – und zwar mithilfe eines menschlichen Modells, das echtes Darmgewebe imitiert. Die Studie zeigt außerdem, dass sich gezüchtete menschliche Mini-Organe zur Untersuchung verschiedener schwerer Infektionen eignen – insbesondere solcher, bei denen bislang fehlende Tiermodelle die Forschung erschwert haben“, ergänzt Maria Letizia Di Martino von der Universität Uppsala, die die Entwicklung des experimentellen Systems leitete.
Darmmodelle aus Stammzellen
Shigella-Bakterien sind invasive Krankheitserreger, die das Körpergewebe mit verschiedenen „Waffen“ angreifen, um in die Darmschleimhaut einzudringen und die Funktionen des körpereigenen Immunsystems zu beeinflussen. In der aktuellen Studie konzentrierten sich die Forschenden darauf, die Gene zu identifizieren, die für die Produktion dieser Waffen verantwortlich sind. Dazu entwickelten sie Darmorganoide, Miniaturmodelle des menschlichen Darms. Sie verwendeten dabei Stammzellen, die aus übrig gebliebenem chirurgischem Gewebe stammten. Mit einer genetischen Methode, bei der einzelne Gene gezielt ausgeschaltet werden, testeten sie, welche Auswirkungen diese Veränderungen auf die Fähigkeit von Shigella haben, das menschliche Darmmodell zu infizieren.
Eine der größten Herausforderungen bei der Anwendung eines solchen Screenings ist das Vorhandensein von Populationsengpässen. Diese Engpässe entstehen zum Beispiel durch Nährstoffmangel oder Konkurrenz um Siedlungsorte. Dadurch sinkt die Zahl der Bakterien vorübergehend – unabhängig davon, wie anpassungsfähig einzelne Mutanten sind. Das führt zu einem zufälligen Verlust von Mutanten und kann somit die Ergebnisse umfangreicher Screenings verfälschen. Um diesen Verlust auszugleichen, hat das Team ein statistisches Modell entwickelt, das Informationen aus einer Vielzahl kleiner Experimente zusammenführt, um eine genomweite Karte zu erstellen. Auf diese Weise gelang es, die erste umfassende Karte der Gene zu erstellen, die Shigella benötigt, um menschliches Darmgewebe zu infizieren.
Besonders überraschend war die Entdeckung, dass bestimmte Veränderungen an Transferribonukleinsäuren (tRNAs, von engl. tranfer ribonucleic acids) die Aktivität des Typ-III-Sekretionssystems steuern können – einem komplexen Apparat, mit dem Bakterien ihre krankmachenden Eigenschaften entfalten. Solche Systeme kosten die Bakterien viel Energie, deshalb steuern sie sie sehr genau. „Bisher war bekannt, dass Bakterien dies beispielsweise durch das Ausschalten fremder DNA oder die Regulierung der Anzahl bestimmter Plasmide – kleiner, ringförmiger DNA-Moleküle – erreichen. Diese Studie zeigt nun einen dritten grundlegenden Mechanismus zur Steuerung auf“, erklärt Laura Jenniches. Sie ist Postdoktorandin im Barquist-Labor und führte die statistische und computergestützte Analyse durch.
„Shigella verfügt über etwa 5.000 Gene, aber wir haben herausgefunden, dass nur etwa 100 davon notwendig sind, um Gewebe zu besiedeln und aggressive Infektionen zu verursachen. Diese Liste ist eine Goldgrube für das Verständnis des Infektionsverlaufs und die Entwicklung neuer Therapien, die das krankmachende Verhalten der Bakterien gezielt ausschalten können“, sagt Mikael Sellin. Er ist Professor an der Universität Uppsala und ein weiterer leitender Autor der Studie. Die eingesetzte Screening-Technologie sei auf viele weitere Erreger übertragbar – und ermögliche künftig realitätsnahe Studien an menschlichen Geweben. Lars Barquist blickt voraus: „Diese Studie schafft die technischen Voraussetzungen für die Untersuchung zahlreicher Krankheitserreger in realistischen Organoidmodellen, die zentrale Aspekte der menschlichen Physiologie im Labor nachbilden.“