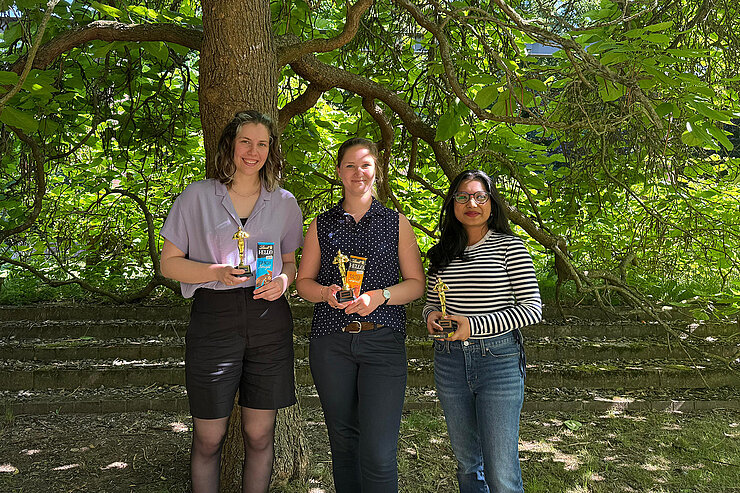
Dynamik respiratorischer Infektionen

Unsere Forschung
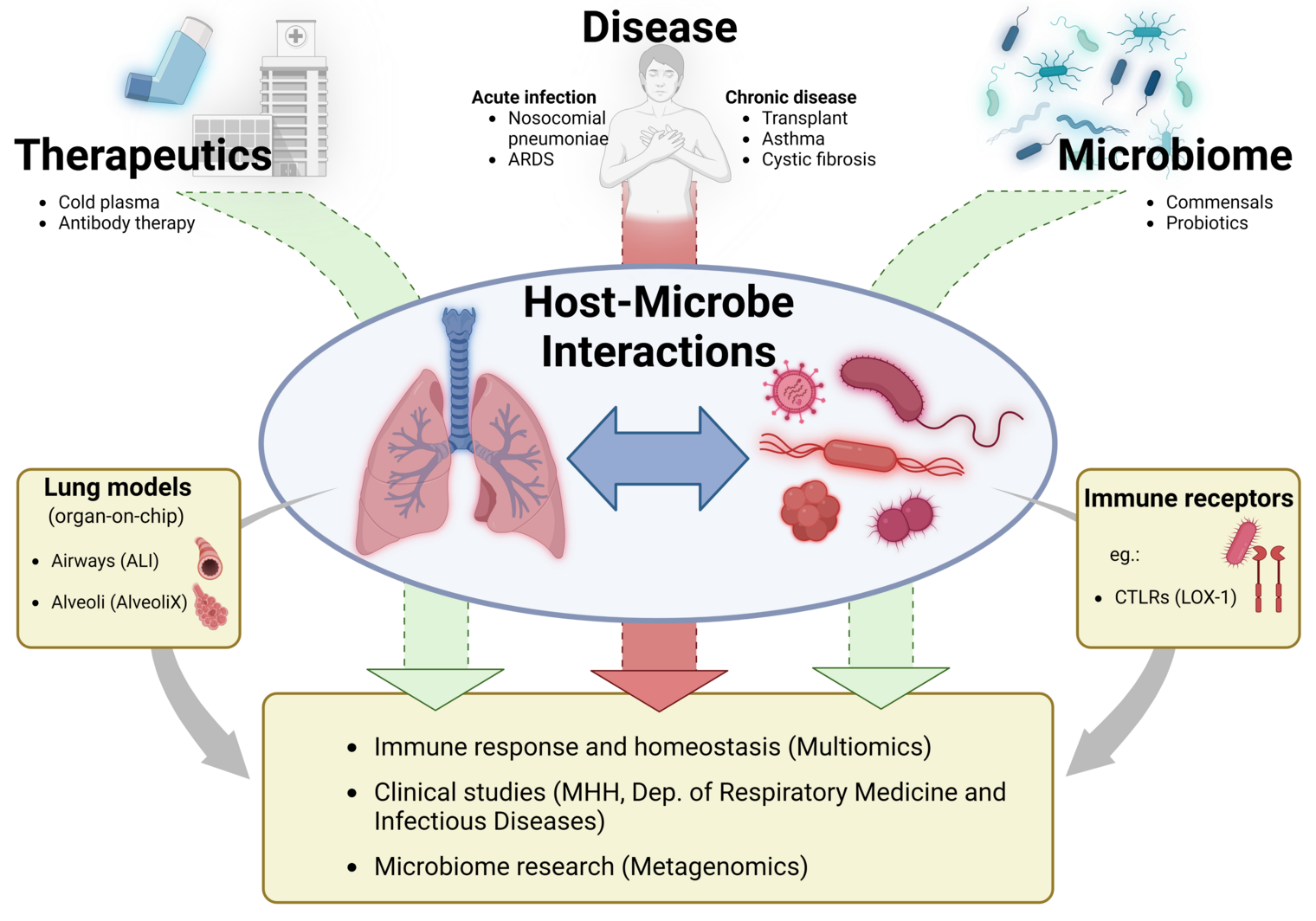
Mikrobiom, Immunantwort und Pathogenese in der Lunge
Das respiratorische Mikrobiom ist ein zentraler, aber bislang unzureichend verstandener Bestandteil der Lungenhomöostase. Es trägt wesentlich zur Aufrechterhaltung des immunologischen Grundtonus, zur Kolonisierungsresistenz gegenüber Pathogenen und zur Kontrolle entzündlicher Prozesse bei. Im Gegensatz zum Darmmikrobiom sind die mikrobiellen Gemeinschaften der unteren Atemwege jedoch bislang kaum charakterisiert – insbesondere in Bezug auf ihre funktionelle Rolle in Gesundheit und Krankheit.
Ein wesentlicher Grund liegt in den methodischen Herausforderungen: Die mikrobielle Biomasse in der Lunge ist gering, die Proben – z. B. aus bronchoalveolärer Lavage – enthalten einen hohen Anteil humaner DNA. Zudem fehlen standardisierte Methoden zur zuverlässigen Analyse nicht-bakterieller Mikroben wie Pilze (Mykobiom) und Viren (Virom). Bis heute mangelt es an vergleichenden Untersuchungen zur Leistungsfähigkeit unterschiedlicher Sequenzieransätze, Primer-Spezifitäten und Protokollen für die Analyse mikrobieller Signaturen aus dem Lungenraum.
Unsere Arbeitsgruppe ist in einzigartiger Weise an der Schnittstelle von klinischer Infektionsmedizin und translationaler Mikrobiomforschung positioniert – als Teil der Klinik für Pneumologie und Infektiologie der Medizinischen Hochschule Hannover (MHH) sowie gleichzeitig als Forschungsgruppe „Dynamik respiratorischer Infektionen (DINF)“ am Helmholtz-Zentrum für Infektionsforschung (HZI) in Braunschweig.
Diese doppelte Verankerung ermöglicht eine unmittelbare Rückkopplung zwischen klinischer Beobachtung und grundlagenwissenschaftlicher Analyse. Durch diese Struktur können wir gezielt Patientenmaterial und klinische Fragestellungen in experimentelle Systeme überführen – und umgekehrt molekulare Erkenntnisse direkt in diagnostische oder therapeutische Anwendungen rückspiegeln. Sie bildet damit ein ideales Modell für eine translationale Infektionsforschung mit Schwerpunkt Lungeninfektionen von der Klinik ins Labor und zurück.
Wir setzen modernste Sequenziertechnologien – darunter ampliconbasierte ITS- und 16S-Sequenzierung, Shotgun-Metagenomik sowie Long-Read-Technologien – ein, um eine möglichst umfassende Erfassung der mikrobiellen Gemeinschaften zu ermöglichen. Ergänzend entwickeln wir bioinformatische Auswertestrategien, um mikrobielle Signaturen auch bei niedriger Biomasse und hohem Anteil humaner DNA sicher identifizieren zu können. Ein besonderer Schwerpunkt liegt auf der methodischen Erschließung der Pilzkomponenten (Mykobiom) des respiratorischen Mikrobioms, deren funktionelle Rolle in mikrobiellen Netzwerken und in der Interaktion mit humanen Zellen bislang weitgehend ungeklärt ist.
Unser Ziel ist es, die mikrobiellen Ökosysteme der Lunge umfassend zu charakterisieren und ihre Rolle in der Pathogenese respiratorischer Erkrankungen – etwa der ambulant oder nosokomial erworbenen Pneumonie – besser zu verstehen. Im Fokus stehen dabei sowohl klassische Pathogene wie Streptococcus pneumoniae, Staphylococcus aureus, Klebsiella pneumoniae und Escherichia coli, als auch bislang wenig untersuchte Kommensalen wie Streptococcus mitis, Prevotella spp., Moraxella catarrhalis und Haemophilus influenzae, die potenziell immunmodulierende Funktionen ausüben. Darüber hinaus analysieren wir die funktionelle Integration fungaler Spezies – insbesondere Candida spp. und Aspergillus spp. – in die mikrobiellen Gemeinschaften der Lunge und deren Wechselwirkungen mit bakteriellen Mikroben und Wirtszellen.
Zur funktionellen Analyse nutzen wir fortschrittliche experimentelle Plattformen, darunter Air-Liquid-Interface (ALI) Kulturen, Lung-on-Chip-Systeme, patientenabgeleitete Lungengewebeschnitte (PCLS) sowie humane Lungenorganoide. Diese Modelle erlauben uns, die Interaktionen zwischen Mikroorganismen, respiratorischem Epithel und alveolären Makrophagen in einem immunologisch relevanten Kontext zu untersuchen.
Langfristig zielt unsere Forschung darauf ab, die mikrobielle Signatur und Dynamik in der Lunge nicht nur besser zu verstehen, sondern auch für die klinische Diagnostik und neue therapeutische Strategien nutzbar zu machen – etwa durch gezielte Mikrobiommodulation, durch die Identifikation diagnostischer Marker oder durch innovative Ansätze zur Prävention und Therapie respiratorischer Infektionen.
Unsere Forschung
Mikrobiom, Immunantwort und Pathogenese in der Lunge
Das respiratorische Mikrobiom ist ein zentraler, aber bislang unzureichend verstandener Bestandteil der Lungenhomöostase. Es trägt wesentlich zur Aufrechterhaltung des immunologischen Grundtonus, zur Kolonisierungsresistenz gegenüber Pathogenen und zur Kontrolle entzündlicher Prozesse bei. Im Gegensatz zum Darmmikrobiom sind die mikrobiellen Gemeinschaften der unteren Atemwege jedoch bislang kaum charakterisiert – insbesondere in Bezug auf ihre funktionelle Rolle in Gesundheit und Krankheit.
Ein wesentlicher Grund liegt in den methodischen Herausforderungen: Die mikrobielle Biomasse in der Lunge ist gering, die Proben – z. B. aus bronchoalveolärer Lavage – enthalten einen hohen Anteil humaner DNA. Zudem fehlen standardisierte Methoden zur zuverlässigen Analyse nicht-bakterieller Mikroben wie Pilze (Mykobiom) und Viren (Virom). Bis heute mangelt es an vergleichenden Untersuchungen zur Leistungsfähigkeit unterschiedlicher Sequenzieransätze, Primer-Spezifitäten und Protokollen für die Analyse mikrobieller Signaturen aus dem Lungenraum.
Unsere Arbeitsgruppe ist in einzigartiger Weise an der Schnittstelle von klinischer Infektionsmedizin und translationaler Mikrobiomforschung positioniert – als Teil der Klinik für Pneumologie und Infektiologie der Medizinischen Hochschule Hannover (MHH) sowie gleichzeitig als Forschungsgruppe „Dynamik respiratorischer Infektionen (DINF)“ am Helmholtz-Zentrum für Infektionsforschung (HZI) in Braunschweig.
Diese doppelte Verankerung ermöglicht eine unmittelbare Rückkopplung zwischen klinischer Beobachtung und grundlagenwissenschaftlicher Analyse. Durch diese Struktur können wir gezielt Patientenmaterial und klinische Fragestellungen in experimentelle Systeme überführen – und umgekehrt molekulare Erkenntnisse direkt in diagnostische oder therapeutische Anwendungen rückspiegeln. Sie bildet damit ein ideales Modell für eine translationale Infektionsforschung mit Schwerpunkt Lungeninfektionen von der Klinik ins Labor und zurück.
Wir setzen modernste Sequenziertechnologien – darunter ampliconbasierte ITS- und 16S-Sequenzierung, Shotgun-Metagenomik sowie Long-Read-Technologien – ein, um eine möglichst umfassende Erfassung der mikrobiellen Gemeinschaften zu ermöglichen. Ergänzend entwickeln wir bioinformatische Auswertestrategien, um mikrobielle Signaturen auch bei niedriger Biomasse und hohem Anteil humaner DNA sicher identifizieren zu können. Ein besonderer Schwerpunkt liegt auf der methodischen Erschließung der Pilzkomponenten (Mykobiom) des respiratorischen Mikrobioms, deren funktionelle Rolle in mikrobiellen Netzwerken und in der Interaktion mit humanen Zellen bislang weitgehend ungeklärt ist.
Unser Ziel ist es, die mikrobiellen Ökosysteme der Lunge umfassend zu charakterisieren und ihre Rolle in der Pathogenese respiratorischer Erkrankungen – etwa der ambulant oder nosokomial erworbenen Pneumonie – besser zu verstehen. Im Fokus stehen dabei sowohl klassische Pathogene wie Streptococcus pneumoniae, Staphylococcus aureus, Klebsiella pneumoniae und Escherichia coli, als auch bislang wenig untersuchte Kommensalen wie Streptococcus mitis, Prevotella spp., Moraxella catarrhalis und Haemophilus influenzae, die potenziell immunmodulierende Funktionen ausüben. Darüber hinaus analysieren wir die funktionelle Integration fungaler Spezies – insbesondere Candida spp. und Aspergillus spp. – in die mikrobiellen Gemeinschaften der Lunge und deren Wechselwirkungen mit bakteriellen Mikroben und Wirtszellen.
Zur funktionellen Analyse nutzen wir fortschrittliche experimentelle Plattformen, darunter Air-Liquid-Interface (ALI) Kulturen, Lung-on-Chip-Systeme, patientenabgeleitete Lungengewebeschnitte (PCLS) sowie humane Lungenorganoide. Diese Modelle erlauben uns, die Interaktionen zwischen Mikroorganismen, respiratorischem Epithel und alveolären Makrophagen in einem immunologisch relevanten Kontext zu untersuchen.
Langfristig zielt unsere Forschung darauf ab, die mikrobielle Signatur und Dynamik in der Lunge nicht nur besser zu verstehen, sondern auch für die klinische Diagnostik und neue therapeutische Strategien nutzbar zu machen – etwa durch gezielte Mikrobiommodulation, durch die Identifikation diagnostischer Marker oder durch innovative Ansätze zur Prävention und Therapie respiratorischer Infektionen.
Prof. Dr. med. Hortense Slevogt
Ein besseres Verständnis der Wirt-Mikroben-Interaktionen in der Lunge könnte uns helfen, neue therapeutische Strategien für eine Vielzahl von Atemwegsinfektionen zu entwickeln.

Prof. Dr. med. Hortense Slevogt ist Internistin und Infektiologin. Seit 2022 ist leitende Oberärztin für klinische Infektiologie an der Klinik für Pneumologie und Infektiologie der Medizinischen Hochschule Hannover (MHH).
Sie hat sich als renommierte Forscherin und Ärztin auf dem Gebiet der Atemwegsinfektionen und Immunologie etabliert. Ihr akademischer Weg begann mit einem Studium der Medizin an der Freien Universität Berlin, während dessen sie ein besonderes Interesse an Infektionskrankheiten und deren Auswirkungen auf die Gesundheit der Atemwege entwickelte. Angetrieben von ihrer Leidenschaft, die komplizierten Abläufe des menschlichen Immunsystems zu verstehen, spezialisierte sie sich weiter auf Innere Medizin und Pulmologie und wurde zu einer anerkannten Expertin auf diesem Gebiet.
Im Jahr 2009 wurden Prof. Slevogts Leistungen durch den Abschluss ihrer Habilitation in Innerer Medizin an der Charité - Universitätsmedizin Berlin gewürdigt. Dies war ein bedeutender Meilenstein, der ihr Fachwissen und ihr tiefes Verständnis für die komplexe Natur schwerer Infektionen unterstrich. Einige Jahre später, im Jahr 2011, übernahm Prof. Slevogt die Leitung der Host Septomics Group am Universitätsklinikum Jena.
Im Jahr 2022 erhielt sie die W3-Professur an der Medizinischen Hochschule Hannover (MHH) und wurde Leiterin der Forschungsgruppe "Dynamik respiratorischer Infektionen" (DINF) am Helmholtz-Zentrum für Infektionsforschung (HZI) in Braunschweig. In dieser Funktion leitet sie translationale Projekte, bei denen sie mit renommierten akademischen und medizinischen Partnern der MHH zusammenarbeitet. Im Rahmen dieser Projekte untersucht Dr. Slevogt das Wirt-Erreger-Interaktom und klärt die Mechanismen der Immunantwort und deren Kontrolle auf.
Parallel zu ihrer Forschungstätigkeit widmet sich Dr. Slevogt der klinischen Praxis, um sicherzustellen, dass ihre Erkenntnisse direkt den Patienten zugutekommen. Als leitende Oberärztin für klinische Infektiologie in der Abteilung für Atemwegsmedizin und Infektionskrankheiten (MHH) trägt sie mit ihrer Expertise zur Diagnose und Behandlung von Atemwegsinfektionen bei.
Team










Auszeichnung für den besten Vortrag in Mikrobiologie für Sarah Truthe auf der ZIB-Klausur 2025
Wir freuen uns, dass unsere Doktorandin Sarah Truthe (in der Mitte) auf der ZIB-Klausur im Juni 2025 mit dem Preis für den besten Vortrag in Mikrobiologie ausgezeichnet wurde. Ihre Präsentation überzeugte die Zuhörer, die ihren Vortrag zum besten in der Sektion Mikrobiologie wählten. Herzlichen Glückwunsch an Sarah zu dieser wohlverdienten Anerkennung ihrer hervorragenden Arbeit!
Teamfoto

Ausgewählte Publikationen
Zubiria-Barrera C), Yamba LY) , Klassert TE, Bos M, Ahl J, Wasserstrom L, Slevogt H,* Riesbeck K* (2025). Profiling the nasopharyngeal Microbiome in patients with community-acquired pneumonia caused by Streptococcus pneumoniae: diagnostic challenges and ecological insights. Med Microbiol Immunol. 2025 Apr 10;214(1):19. doi: 10.1007/s00430-025-00828-0.
Klassert TE, Zubiria-Barrera C, Denkel L, Neubert R, Schneegans A, Kulle A, Vester A, Bloos F, Schulze C, Epstude J, Gastmeier P, Geffers C, Slevogt H (2024). Skin dysbiosis and loss of microbiome site specificity in critically ill patients. Microbiol Spectr. 2024 Mar 5;12(3):e0307823. doi: 10.1128/spectrum.03078-23.
Müller MM, Baldauf C, Hornischer S, Klassert TE, Schneegans A, Behnert A, Pletz MW, Hagel S, Slevogt H §. (2023). Staphylococcus aureus induces tolerance in human monocytes accompanied with expression changes of cell surface markers. Front Immunol. 14:1046374. doi: 10.3389/fimmu.2023.1046374.
Klassert TE , Leistner R, Zubiria-Barrera C, Stock M, López M, Neubert R, Driesch D, Gastmeier P, Slevogt H §. (2021). Bacterial colonization dynamics and antibiotic resistance gene dissemination in the hospital environment after first patient occupancy: a longitudinal metagenetic study. Microbiome. 9(1):169. doi: 10.1186/s40168-021-01109-7.
Lehmann R, Müller MM, Klassert TE, Driesch D, Stock M, Heinrich A, Conrad T, Moore C, Schier UK, Guthke R, Slevogt H§. (2018). Differential regulation of the transcriptomic and secretomic landscape of sensor and effector functions of human airway epithelial cells. Mucosal Immunol. 11(3):627-642. doi: 10.1038/mi.2017.100.
Projekte
- PlasmaCare: Sicherheitsanalyse der Anwendung von kaltem Plasma auf das menschliche Lungenepithel in komplexen Zellkulturmodellen Charakterisierung des respiratorischen Mikrobioms bei Gesunden und bei Patienten mit ventilatorassoziierter Pneumonie
- AsthmaBiom: Veränderungen des respiratorischen Mikrobioms bei Patienten mit schwerem Asthma vor und nach immunmodulatorischer Antikörpertherapie und deren Assoziation mit klinischen Verlaufsparametern
- Der Einfluss mikrobieller Kommensalen-Pathogen-Interaktionen auf die Gestaltung des menschlichen Respirationstrakts (RESIST)
- Das Zusammenspiel von Candida albicans- und Aspergillus fumigatus-induzierten Signalen bei der rezeptorvermittelten Modulation der antimykotischen Immunantwort
- Entwicklung von Point-of-Care-Tests (POCT) für die Diagnostik von Infektionen mit Pneumozystis jirovecii und Aspergillus fumigatus