In Ihrer neuen Publikation geht es um den Naturstoff Darobactin. Wo kommt dieses Molekül her und warum ist es für Sie so interessant?
Darobactin ist ein Antibiotikum, das erstmals 2019 von Imai und Kollegen in der Fachzeitschrift Nature beschrieben wurde und natürlicherweise vom Bakterium Photorhabdus khanii HGB1456 produziert wird. Dieses Bakterium lebt in Symbiose mit Nematoden, also Fadenwürmern, die unter anderem Insektenlarven befallen und sich von diesen ernähren. Die Fähigkeit von P. khanii, Darobactin zu produzieren, gibt den Nematoden einen entscheidenden Vorteil, denn das Antibiotikum tötet schädliche Bakterien, die beispielsweise aus den befallenen Insektenlarven stammen. Ihre eigenen, für die Nematoden wichtigen Darmbakterien sind hingegen immun gegen Darobactin. Basierend auf der Analyse von Genomdaten wurde außerdem vorhergesagt, dass Darobactin auch von einer Vielzahl weiterer Bakterien produziert werden könnte - unter anderem von Yersinia pestis, dem Erreger der schwarzen Pest.
Darobactin ist interessant für uns, weil es als Antibiotikum spezifisch gegen gramnegative Bakterien wirkt. Diese Bakterien besitzen im Gegensatz zu grampositiven Bakterien eine doppelte Zellmembran, welche eine schwer überwindbare Barriere für viele Antibiotika darstellt. Daher ist es nicht verwunderlich, dass viele der multiresistenten und schwer zu behandelnden Krankenhauskeime gramnegativ sind. Es ist wichtig für uns, neue Antibiotika-Strukturen zu entdecken und weiterzuentwickeln, um auch zukünftig weiterhin wirksame Behandlungsoptionen für bakterielle Infektionen zu haben. Darobactin zeigte in bisherigen Tests nicht nur eine außergewöhnlich starke antimikrobielle Aktivität gegen viele für den Menschen gefährliche gramnegative Bakterien, sondern es verschont auch hilfreiche Bakterien, die im Darm des Menschen vorkommen und wichtig für die Verdauung sind. Einige der heutzutage auf dem Markt befindlichen Antibiotika haben die unangenehme Nebenwirkung, dass sie auch nützliche Bakterien im Darm angreifen, wohingegen dieses Problem bei der Verabreichung von Darobactin möglicherweise nicht auftreten würde.
Wie kann es denn sein, dass Darobactin nur die gramnegativen Bakterien bekämpft?
Die Spezifität gegenüber gramnegativen Bakterien ist darauf zurückzuführen, dass Darobactin an ein spezielles Protein in der äußeren Zellmembran des Bakteriums bindet und dessen Funktion hemmt. Da ausschließlich gramnegative Bakterien diese äußere Zellmembran besitzen, werden auch nur diese durch Darobactin bekämpft. Das gehemmte Protein heißt BamA und ist normalerweise am Aufbau der äußeren Zellmembran beteiligt. Wenn diese Funktion wegfällt, sind die Bakterien nicht mehr überlebensfähig. Es gibt bisher übrigens noch kein anderes Antibiotikum, das in der Humanmedizin eingesetzt wird und BamA als molekulares Ziel hat.
Warum ist es so wichtig, dass das molekulare Ziel von Darobactin bisher noch von keinem anderen Antibiotikum anvisiert wird?
Das hängt mit dem Mechanismus zusammen, wie Bakterien Resistenzen gegen die eingesetzten Antibiotika entwickeln. Bakterien haben im Laufe der Evolution verschiedene Strategien entwickelt, die Angriffsfläche, die sie Antibiotika bieten, zu verringern. So können sie beispielsweise vermehrt Zellmembrantransporter produzieren, um das Antibiotikum so effizient aus der Zelle heraus zu transportieren, dass es sein Ziel in der Zelle nicht mehr erreichen kann. Eine andere Möglichkeit wäre die enzymatische Modifikation des Antibiotikums selbst. Das kann entweder durch eine Spaltung des Antibiotikums passieren, wie z.B. bei Penicillin, oder durch das Anhängen funktioneller Gruppen wie Phosphatgruppen bei Aminoglykosid-Antibiotika. Dadurch verliert das jeweilige Antibiotikum seine antimikrobielle Wirkung. Eine weitere Art der Resistenzentwicklung ist eine Veränderung der Struktur des molekularen Ziels durch zufällige Mutationen, sodass das Antibiotikum schlichtweg nicht mehr an sein Ziel binden kann und dadurch seine Wirkung verliert. Da es einige Antibiotikaklassen in der Humanmedizin gibt, die das gleiche molekulare Ziel im Bakterium haben, verlieren diese dann unter Umständen alle zugleich ihre Wirksamkeit. Wir sprechen dann von einer sogenannten Kreuzresistenz. Dieses Problem kann aber durch Antibiotika umgangen werden, die ein völlig neues Ziel haben, wie BamA im Falle von Darobactin.
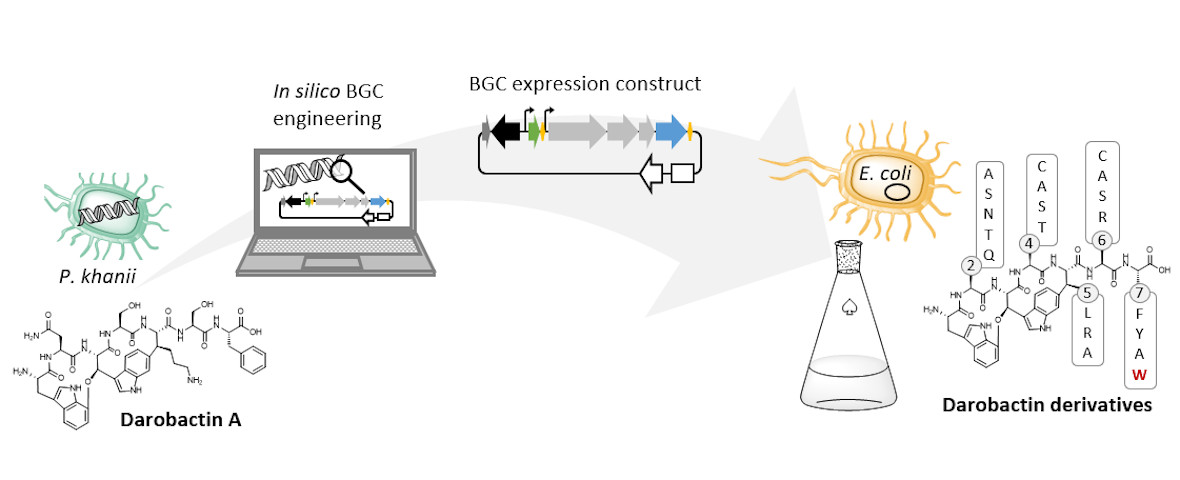
In Ihrer Studie haben Sie eine Strategie entwickelt, um die Produktion von Darobactin in größeren Mengen zu ermöglichen. Warum ist das notwendig und wie sind Sie dabei vorgegangen?
Der natürliche Produzent von Darobactin ist ein weniger gut charakterisiertes Bakterium, das im Labor nur sehr wenig Darobactin produziert. Zur genaueren Charakterisierung und der präklinischen Entwicklung dieser Substanzklasse werden jedoch größere Stoffmengen benötigt, als bislang durch die vorhandenen Prozesse mit dem natürlichen Produzenten bereitgestellt werden konnten. Da es kaum etablierte Protokolle zur schnellen Produktionsverbesserung mit dem natürlichen Produzentenstamm gibt und die Entwicklung dieser Methoden sehr viel Zeit und Arbeitsaufwand in Anspruch nehmen würde, haben wir uns entschieden, auf Escherichia coli als alternativen Produzenten auszuweichen. E. coli ist eines der am besten charakterisierten Bakterien, produziert natürlicherweise aber kein Darobactin. Deshalb haben wir alle Gene, die der natürliche Produzent zur Bildung von Darobactin benötigt, in E. coli übertragen. Den Aufbau des biosynthetischen Genclusters (BGC) haben wir vorher virtuell auf eine Expression in E. coli angepasst, um am Ende eine möglichst hohe Darobactin-Ausbeute erreichen zu können. Das BGC wurde dann mittels DNA-Synthese hergestellt, in einen Expressionsvektor kloniert und in E. coli eingebracht. Der erzeugte E. coli-Stamm konnte tatsächlich das vorher von Imai und Kollegen beschriebene Derivat Darobactin A heterolog produzieren - allerdings mit einer über vierfach erhöhten Produktionsrate verglichen zum natürlichen Produzenten.
Bei der Expression des Darobactin-BGCs haben Sie neben dem erwarteten Darobactin A auch noch 17 neue Derivate entdecken können. War etwas Spannendes darunter? Konnten Sie aus diesen Ergebnissen neue Erkenntnisse ziehen?
Die 17 neuen Derivate haben wir nicht zufällig entdeckt, sondern bewusst darauf abgezielt, diese zu produzieren. Wir haben dabei gezielt eine Gensequenz im Darobactin-BGC verändert, um die Produktion dieser Derivate in E. coli zu ermöglichen. Zunächst haben wir grob die antimikrobielle Aktivität der verschiedenen Derivate gegen eine kleine Anzahl gramnegativer Pathogene abgeschätzt, um zu sehen, welche Derivate überhaupt noch eine antibakterielle Aktivität aufweisen. Nicht selten verlieren Antibiotika bei struktureller Modifikation nämlich ihre Aktivität. Bei den von uns untersuchten Darobactin-Derivaten gab es allerdings einige vielversprechende Kandidaten. Aufgrund des aufwändigen Darobactin-Aufreinigungsprozesses aus der Fermentationskultur haben wir uns vorerst nur auf das Derivat Darobactin 9 fokussiert und dieses isoliert. Im Vergleich zu Darobactin A zeigte dieses Derivat glücklicherweise sogar eine verbesserte antimikrobielle Aktivität gegenüber einigen wichtigen Krankenhauskeimen, insbesondere gegen verschiedene Pseudomonas aeruginosa- und Acinetobacter baumannii-Stämme, bei denen heute keine effizienten Behandlungsmethoden mehr existieren. Während das Aktivitätsprofil von Darobactin A gegen diese Stämme noch Lücken aufwies, kommen wir hier mit Darobactin 9 schon einen bedeutenden Schritt näher zur Anwendbarkeit am Menschen. Natürlich sind bis dahin noch eine Vielzahl an Experimenten im Rahmen (prä-)klinischer Studien nötig.
Wie geht es jetzt weiter? Was sind die nächsten Schritte?
Wir haben uns zum Ziel gesetzt, die Produktionsausbeute weiter zu verbessern. Bisher haben wir die Fermentation unserer E. coli-Produktionsstämme nur in Schüttelkolben durchgeführt. Die Kultivierung unter kontrollierten und optimierten Bedingungen in einem Fermenter größeren Maßstabs birgt hier noch großes Verbesserungspotenzial.
Darüber hinaus planen wir einerseits die strukturelle Vielfalt der Darobactine noch zu erweitern, indem wir weitere Darobactin-Derivate produzieren, und andererseits einige der 17 bereits produzierten Darobactin-Derivate mit vielversprechender antimikrobieller Aktivität aufzureinigen und tiefgehend zu charakterisieren.
Das Interview führten Dr. Alwin Hartman und Dr. Yannic Nonnenmacher.
Originalpublikation:
Sebastian Groß, Fabian Panter, Domen Pogorevc, Carsten E. Seyfert, Selina Deckarm, Chantal D. Bader, Jennifer Herrmann, Rolf Müller: Improved broad-spectrum antibiotics against Gram-negative pathogens via darobactin biosynthetic pathway engineering. Chem. Sci. 2021; doi: 10.1039/D1SC02725E