Bakteriophagen - Viren, die Bakterien befallen
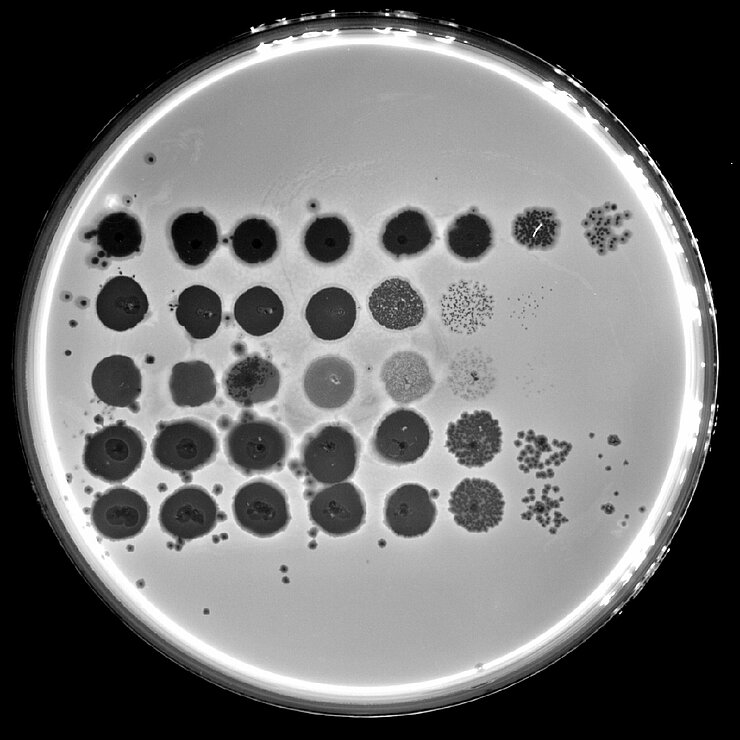
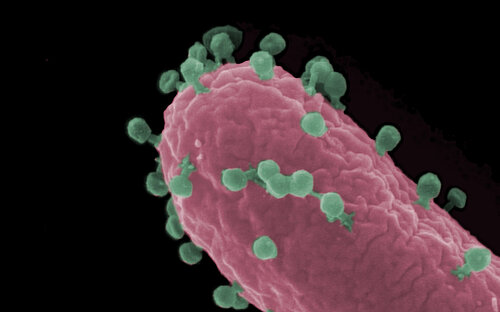
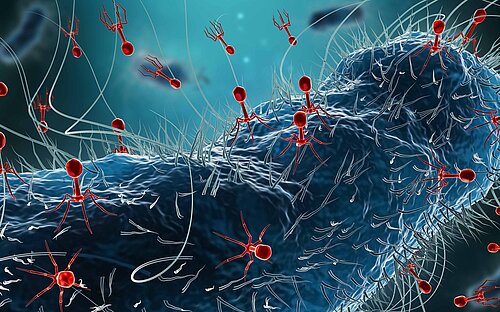
Der Feind meines Feindes ist mein Freund – dieser Satz könnte so auch auf Bakteriophagen zutreffen. Bakteriophagen (kurz: Phagen) sind Viren, die Bakterien befallen und somit deren natürliche Feinde. Ihrem vom griechischen Wort für „Bakterienfresser“ abgeleiteten Namen machen sie alle Ehre: Bakteriophagen docken an Bakterien an, schleusen ihr Erbgut ein und programmieren die Zellen so um, dass diese große Mengen Phagen-Erbgut produzieren. Schließlich platzen die Zellen und setzen neue Phagen frei. Diese können wie bei einer Kettenreaktion wiederum weitere Bakterienzellen infizieren. So können Bakteriophagen beispielsweise in einen dichten Zellrasen mit Bakterien Löcher „fressen“. Allerdings befallen Bakteriophagen nicht wahllos Bakterien, sondern sie sind hochspezialisiert auf eine Wirtsspezies oder sogar nur einzelne Stämme dieser Spezies.
Bakteriophagen sind mit einer Größe von 30 bis 150 Nanometern zwar sehr klein und damit mit einem Lichtmikroskop nicht sichtbar, aber allgegenwärtig. Keine andere biologische Einheit (denn Lebewesen sind Phagen strenggenommen nicht) kommt weltweit häufiger vor. Dadurch spielen Phagen eine wichtige Rolle in allen Ökosystemen. Studien haben ergeben, dass sich in einem Milliliter Meerwasser bis zu zehn Millionen Bakteriophagen befinden und es wird geschätzt, dass diese täglich etwa ein Fünftel aller Bakterien in den Ozeanen töten. Dies setzt Nährstoffe für andere Spezies frei und hält das Ökosystem im Gleichgewicht. Bakteriophagen sind in allen Lebensräumen zu finden, in denen ihre Wirte vorkommen. Daher suchen Wissenschaftler:innen insbesondere im Abwasser großer Städte und von Kliniken nach neuen Bakteriophagen, die Krankheitserreger bekämpfen können, darunter auch solche, die häufig in Krankenhäusern vorkommen.
Phagentherapie: Chancen und Hürden
Die Fähigkeit, Bakterienpopulationen effizient zu dezimieren, macht Bakteriophagen zu einer attraktiven therapeutischen Alternative gegen Infektionen mit multiresistenten Keimen. Seit der Entdeckung der Antibiotika wurde die Phagentherapie jedoch nur wenig erforscht. Aufgrund der zunehmenden Ausbreitung von Antibiotikaresistenzen ist sie wieder mehr in den Fokus gerückt. Bei der Phagentherapie wird bei schweren Infektionen zunächst der Erreger bestimmt und anschließend ein oder mehrere passende Phagen verabreicht, die gegen diesen Erreger wirksam sind. In klinischen Einzelfällen ist dies auch bereits erfolgreich durchgeführt worden. Vor einer Massenanwendung stehen aber hohe wissenschaftliche und regulatorische Hürden. So muss für jede Infektion eine individuelle Rezeptur hergestellt werden, was nach aktuellen Maßstäben für klinische Studien und Zulassungen nicht umsetzbar ist. Um dies in Zukunft zu ermöglichen, ermitteln die Weltgesundheitsorganisation (WHO) und der Global Antimicrobial Resistance Research and Development Hub (Global AMR R&D Hub) gemeinsam die regulatorischen und gesetzlichen Anforderungen, um den breiteren Einsatz von Phagen zur Bekämpfung antimikrobieller Resistenzen zu unterstützen.
Ein Werkzeugkasten für die Forschung
Bakterien sind den Angriffen durch Bakteriophagen nicht wehrlos ausgeliefert. Im Laufe von vielen Millionen Jahren der Evolution haben Bakterien zahlreiche Systeme entwickelt, um Phageninfektionen abzuwehren. Das bekannteste bakterielle Immunsystem ist das CRISPR-Cas-System, mit dem Bakterien DNA-Abschnitte von Bakteriophagen erkennen und zerschneiden können – so wird die Infektion verhindert. Forscher:innen haben daraus eine Genschere entwickelt, die zielgenau Gensequenzen in menschlichen Zellen erkennen, schneiden und bearbeiten kann.
In den Genomen von Bakteriophagen schlummern aber noch viele Gene, deren Funktionen bislang völlig unbekannt sind. Genau diese könnten allerdings wichtige Quellen für die Entdeckung neuer molekularer Werkzeuge darstellen. Deshalb untersuchen Forscher:innen am Helmholtz-Zentrum für Infektionsforschung (HZI) und am Helmholtz-Institut für RNA-basierte Infektionsforschung (HIRI) die Wirkweisen dieser Gene, indem sie deren zugrundeliegenden Mechanismen in Phagen-Wirt-Interaktionen aufdecken. Sie wollen insbesondere besser verstehen, wie Bakteriophagen die Kontrolle über ihre Wirte übernehmen, um sich zu vermehren. Hierzu erforschen die HZI- und HIRI-Wissenschaftler:innen beispielsweise Jumbophagen, die über mehrere hundert Gene in ihrem Erbgut verfügen, deren Aufgaben größtenteils unbekannt sind. Am HIRI, das sich auf die Erforschung der Rolle von RNA bei Infektionen konzentriert, studieren Wissenschaftler:innen auch RNA-Phagen. Das Erbgut dieser Phagen ist ausgesprochen klein und besteht – anders als bei den meisten Phagen, die ein aus DNA bestehendes Erbgut haben – aus RNA. RNA-Phagen haben bis dato wenig Beachtung in der Forschung gefunden. Mit der Einführung neuer Sequenzierungstechnologien hat sich das jedoch geändert: Es ist nun möglich, diese Phagen in der Umwelt aufzuspüren. Dadurch hat sich unser Wissen über diese wenig erforschte Gruppe von Phagen erheblich erweitert.
Bakteriophagen sind mehr als nur natürliche Feinde von Bakterien: Sie sind zentrale Akteure in nahezu allen Ökosystemen, eine vielversprechende Quelle für neue biotechnologische Instrumente und ein Hoffnungsträger im Kampf gegen multiresistente Keime. Aktuelle Forschungsprojekte am HZI vertiefen unser Grundverständnis von Virus‑Wirt‑Interaktionen und liefern neue biotechnologische Werkzeuge. Die nächsten Jahre werden zeigen, ob und wie diese Erkenntnisse den Weg zu breit einsetzbaren, sicheren Phagenpräparaten ebnen – ein entscheidender Schritt, um die wachsende Bedrohung durch Antibiotika‑Resistenzen zu meistern.
(cwe)
Stand: November 2025
Beteiligte Forschungsgruppen
-
Evolutionäre Gemeinschaftsökologie
 Dr. Jan Frederik Gogarten
Dr. Jan Frederik Gogarten -
Komplexe in Phageninfizierten Zellen
 Dr. Milan Gerovac
Dr. Milan Gerovac -
Molekulare Grundlagen von RNA-Phagen
 Jun.-Prof. Dr. Jens Hör
Jun.-Prof. Dr. Jens Hör -
RNA-Biologie von bakteriellen Infektionen
 Prof. Dr. Jörg Vogel
Prof. Dr. Jörg Vogel