Biologische Codes von Pathogenen

Unsere Forschung
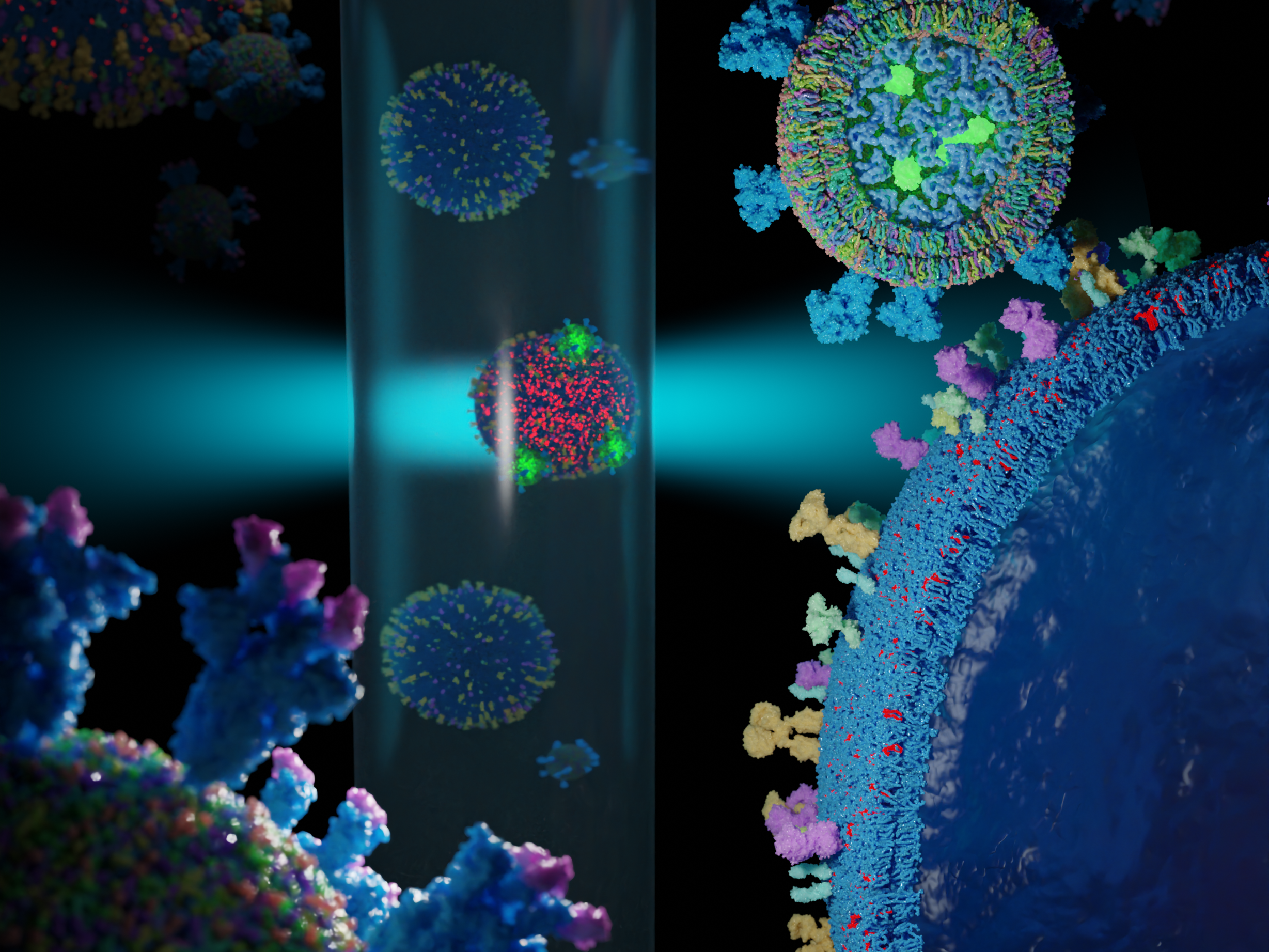
Unsere Forschung konzentriert sich darauf die biologischen Codes von Pathogenen auf molekularer Ebene zu entschlüsseln und beeinflussen zu können. Wenn wir an ein Pathogen denken, zum Beispiel ein einzelnes Virus-Partikel, dann haben wir meist ein vereinfachtes Bild vor Augen wie dieses aussieht und aufgebaut ist. Allerdings vernachlässigen wir dabei wie unterschiedlich dieses einzelne Partikel - selbst bei identischer genetischer Ausstattung - aufgebaut sein kann. Da diese unterschiedlichen Erscheinungsformen Einfluss auf die Eigenschaften des Pathogens nehmen, ist es wichtig, diese besser zu verstehen, um Infektionskrankheiten einzudämmen. Die Vielfalt dieser Erscheinungsformen ist bedingt durch das Zusammenspiel unterschiedlicher biologischer Codes. Unser Team hat es sich zur Aufgabe gesetzt, neue Technologien zur Entschlüsselung dieser Codes zu entwickeln, um deren physiologische Relevanz zu erforschen und sie beeinflussen zu können. Die Erforschung dieser molekularen Codes wird insbesondere durch zwei Hürden erschwert:
- Die nanoskopische Größe vieler Pathogene
- Die Komplexität der Lebenszyklen und Interaktionen mit ihrer Umgebung
Die erste Hürde wird oft durch Signalverstärkung durch das gleichzeitige Messen vieler Pathogene („bulk“) gelöst, wodurch jedoch die Information über die Heterogenität verloren geht. Durch den Einsatz von Hochdurchsatz-Technologien mit Einzelpartikel-Auflösung und fortschrittlichen Mikroskopie-Methoden können wir diese Vielfalt detektieren und analysieren.
Um die komplexen Interaktionen der Pathogene mit ihrer Umgebung besser verstehen zu können, verwenden wir Methoden der synthetischen Biologie, welche es uns erlauben, gewisse Fragestellungen in einem definierten und vereinfachten System zu untersuchen. So können wir zum Beispiel eine stark vereinfachte künstliche Zelle bauen, um das Bindeverhalten von Viren zu analysieren. Durch den Einsatz von umgebungssensitiven Reporter-Molekülen erforschen wir außerdem die kollektiven biophysikalischen Eigenschaften von Pathogenen.
Unsere Forschung
Unsere Forschung konzentriert sich darauf die biologischen Codes von Pathogenen auf molekularer Ebene zu entschlüsseln und beeinflussen zu können. Wenn wir an ein Pathogen denken, zum Beispiel ein einzelnes Virus-Partikel, dann haben wir meist ein vereinfachtes Bild vor Augen wie dieses aussieht und aufgebaut ist. Allerdings vernachlässigen wir dabei wie unterschiedlich dieses einzelne Partikel - selbst bei identischer genetischer Ausstattung - aufgebaut sein kann. Da diese unterschiedlichen Erscheinungsformen Einfluss auf die Eigenschaften des Pathogens nehmen, ist es wichtig, diese besser zu verstehen, um Infektionskrankheiten einzudämmen. Die Vielfalt dieser Erscheinungsformen ist bedingt durch das Zusammenspiel unterschiedlicher biologischer Codes. Unser Team hat es sich zur Aufgabe gesetzt, neue Technologien zur Entschlüsselung dieser Codes zu entwickeln, um deren physiologische Relevanz zu erforschen und sie beeinflussen zu können. Die Erforschung dieser molekularen Codes wird insbesondere durch zwei Hürden erschwert:
- Die nanoskopische Größe vieler Pathogene
- Die Komplexität der Lebenszyklen und Interaktionen mit ihrer Umgebung
Die erste Hürde wird oft durch Signalverstärkung durch das gleichzeitige Messen vieler Pathogene („bulk“) gelöst, wodurch jedoch die Information über die Heterogenität verloren geht. Durch den Einsatz von Hochdurchsatz-Technologien mit Einzelpartikel-Auflösung und fortschrittlichen Mikroskopie-Methoden können wir diese Vielfalt detektieren und analysieren.
Um die komplexen Interaktionen der Pathogene mit ihrer Umgebung besser verstehen zu können, verwenden wir Methoden der synthetischen Biologie, welche es uns erlauben, gewisse Fragestellungen in einem definierten und vereinfachten System zu untersuchen. So können wir zum Beispiel eine stark vereinfachte künstliche Zelle bauen, um das Bindeverhalten von Viren zu analysieren. Durch den Einsatz von umgebungssensitiven Reporter-Molekülen erforschen wir außerdem die kollektiven biophysikalischen Eigenschaften von Pathogenen.
Dr. Jan Schlegel
Pathogene zu verstehen, erfordert mehr als Wissen – es erfordert Vorstellungskraft.

Angetrieben von der Frage, wie das Leben auf molekularer Ebene funktioniert, studierte Jan Schlegel Biologie an der JMU Würzburg. Hier entwickelte er eine besondere Faszination für biophysikalische Methoden und der Enthüllung der verborgenen „Nano-Welt“. Deswegen entschied er sich für ein Promotionsstudium der Lebenswissenschaften in der Arbeitsgruppe von Prof. Markus Sauer, um Methoden der hochaufgelösten Einzelmolekül-Mikroskopie für Lipide im Kontext der Wirt-Pathogen-Interaktion weiterzuentwickeln. Nach dem erfolgreichen Abschluss seiner Doktorarbeit (summa cum laude) wechselte er 2021 als Postdoktorand an das Karolinska Institutet (Prof. Erdinc Sezgin) in Schweden, um mit Hilfe fortschrittlicher Mikroskopie-Methoden und synthetischer Biologie innovative Hochdurchsatz-Screeningverfahren für Nanopartikel zu entwickeln. Die Auszeichnung mit einem kompetitiven Marie Skłodowska-Curie Postdoc-Forschungsstipendium erlaubte es ihm von 2022 bis 2024 durch seine eigene Forschung im Bereich der asymmetrischen Verteilung von Lipiden in biologischen Membranen Akzente zu setzen. Im Anschluss daran wechselte er als Postdoktorand zur Königlich Technischen Hochschule (KTH) in Schweden (Prof. Hjalmar Brismar & Dr. Hans Blom), wo er seine Expertise der Einzelmolekül-Lokalisierungs Mikroskopie zur Beantwortung zellbiologischer Fragestellungen mittels MINFLUX Mikroskopie weiter ausbauen konnte. Seit 01. August 2025 leitet Jan Schlegel die HUMAN (Human Microbe Alliance for Universal Health) Nachwuchsgruppe „Biologische Codes von Pathogenen“ am Helmholtz-Zentrum für Infektionsforschung (HZI) in Braunschweig. Seine Gruppe widmet sich der Erforschung von biologischen Codes (z.B. Glycocode, Biophysikalischer Code, etc.) von Pathogenen, um innovative diagnostische und therapeutische Ansätze gegen Infektionskrankheiten zu entwickeln.
Team




Ausgewählte Publikationen
Milka Doktorova, Jessica L. Symons, Xiaoxuan Zhang, Hong-Yin Wang, Jan Schlegel, Joseph H. Lorent, Frederick A. Heberle, Erdinc Sezgin, Edward Lyman, Kandice R. Levental, Ilya Levental: "Cell Membranes Sustain Phospholipid Imbalance Via Cholesterol Asymmetry" Cell. 2025, DOI: 10.1016/j.cell.2025.02.034
Pablo Carravilla *, Luca Andronico *, Jan Schlegel *, Yagmur B. Urem, Ellen Sjule, Franziska Ragaller, Florian Weber, Cenk O. Gurdap, Yavuz Ascioglu, Taras Sych, Joseph Lorent, Erdinc Sezgin: "Measuring plasma membrane fluidity using confocal microscopy" Nature Protocols. 2025, DOI: 10.1038/s41596-024-01122-8
* equal contribution
Taras Sych, Jan Schlegel, Hanna MG Barriga, Miina Ojansivu, Leo Hanke, Florian Weber, R Beklem Bostancioglu, Kariem Ezzat, Herbert Stangl, Birgit Plochberger, Jurga Laurencikiene, Samir El Andaloussi, Daniel Fürth, Molly M Stevens, Erdinc Sezgin: "High-throughput measurement of the content and properties of nano-sized bioparticles with single-particle profiler" Nature Biotechnology. 2023, DOI: 10.1038/s41587-023-01825-5
Jan Schlegel, Bartlomiej Porebski, Luca Andronico, Leo Hanke, Steven Edwards, Hjalmar Brismar, Ben Murrell, Gerald M. McInerney, Oscar Fernandez-Capetillo, and Erdinc Sezgin: “A Multiparametric and High-Throughput Platform for Host–Virus Binding Screens” Nano Letters. 2023, DOI: 10.1021/acs.nanolett.2c04884
Felix Wäldchen *, Jan Schlegel *, Ralph Götz, Michael Luciano, Martin Schnermann, Sören Doose, Markus Sauer: “Whole-cell imaging of plasma membrane receptors by 3D lattice light-sheet dSTORM” Nature Communications. 2020 * equal contribution DOI: 10.1038/s41467-020-14731-0
Publikationen
Eine vollständige Liste der Publikationen finden Sie hier.

