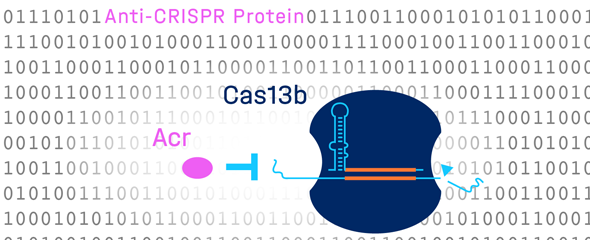
Bakterien enthalten häufig CRISPR-Cas-Abwehrsysteme, die sie vor viralen Eindringlingen, sogenannten Phagen, schützen. Als Reaktion darauf haben Phagen ihrerseits Proteine entwickelt, die diese bakterielle „Genschere“ inaktivieren können. Forschende vermuten, dass es zahlreiche solcher Anti-CRISPR-Proteine (Acrs) gibt, die noch nicht entdeckt wurden. Einer dieser Wissenschaftler ist Chase Beisel, Leiter der Abteilung „Synthetische RNA-Biologie“ am Helmholtz-Institut für RNA-basierte Infektionsforschung (HIRI) in Würzburg, eines Standorts des Braunschweiger Helmholtz-Zentrums für Infektionsforschung (HZI) in Kooperation mit der Julius-Maximilians-Universität (JMU) Würzburg. „Acrs können zur besseren Kontrolle von CRISPR-Technologien eingesetzt werden“, sagt Beisel und räumt zugleich ein: „Sie zu identifizieren heißt, eine Stecknadel im Heuhaufen zu finden, da Acrs einander überhaupt nicht ähneln.“ Dies habe dazu geführt, dass wissenschaftliche Untersuchungen in der Vergangenheit mit neuen Vorhersagemethoden aufwarteten. Der Erfolg dieser Methoden sei jedoch begrenzt, meint Beisel. Er hat die heute im Fachmagazin „Molecular Cell“ veröffentlichte Studie initiiert.
Auf der Suche nach Acrs: Erstmals Deep Learning angewandt
Entsprechend hat ein Team von Wissenschaftler:innen des Würzburger HIRI zusammen mit Spezialisten aus der biologischen Signalforschung an der Universität Freiburg sowie der Informatik an der King Fahd Universität in Saudi-Arabien den Einsatz künstlicher Intelligenz vorangetrieben, um neue Acrs auszumachen.
„Es ist uns mit der Kombination aus komplexem computergestützten Lernen und dem Einsatz eines Hochdurchsatz-Screens gelungen, ein neues Anti-CRISPR-Protein zu entdecken, das die medizinisch relevante Nuklease Cas13b hemmt“, freut sich Katharina Wandera. Die Doktorandin im Labor von Chase Beisel forscht an der Charakterisierung von Acrs und ist eine Erstautorin der vorgelegten Studie.
„Die Millionen von Vorhersagen, die unser Algorithmus macht, eröffnen der Forschung ganz neue Erkenntnismöglichkeiten, ist sich Rolf Backofen von der Universität Freiburg sicher. Seine Gruppe hat den DeepAcr genannten Rechenvorgang für die Studie entwickelt.
Dass mit DeepAcr nicht nur neue Nukleaseblocker entdeckt werden können, die CRISPR-Cas-Systeme hemmen, denen bekannte Acrs fehlen, legt die Studie ebenfalls nahe. „Das identifizierte Anti-CRISPR-Protein AcrVIB1 war bis dato nicht nur unbekannt, es scheint auch einen völlig neuen Wirkmechanismus zu haben“, resümiert Omer Alkhnbashi, Assistenzprofessor an der King Fahd Universität und mit Wandera im Erstautorenduo.
Diesen Wirkmechanismus zu beschreiben, ist einer der nächsten Schritte, die sich nun an die wissenschaftliche Arbeit anschließen sollen.
Originalpublikation
Anti-CRISPR prediction using deep learning reveals an inhibitor of Cas13b nucleases. Wandera KG, Alkhnbashi OS, Bassett HVI, Mitrofanov A, Hauns S, Migur A, Backofen R, Beisel CL (2022). Molecular Cell. DOI: 10.1016/j.molcel.2022.05.003
Die Studie wurde aus Mitteln der Deutschen Forschungsgemeinschaft (DFG) und im Safe Genes Program der Defense Advanced Research Projects Agency (DARPA) gefördert.