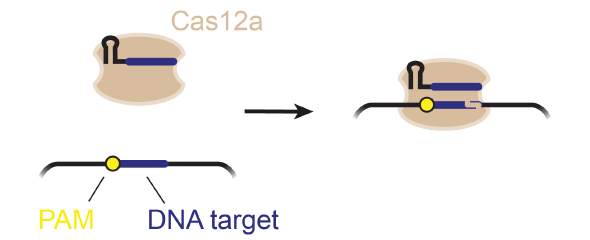
„Das Vorliegen eines geeigneten PAM um die Zielsequenz herum stellt nach wie vor ein Hindernis für die gezielte Genbearbeitung dar“, sagt Prof. Chase Beisel, Leiter der HIRI-Forschungsgruppe „Biologie Synthetischer RNA“. Daher durchforsten Forscher die bestehende Vielfalt von Cas-Nukleasen in Bakterien und Archaeen nach solchen, die neue und einzigartige PAMs erkennen. In Kooperation mit Forschern der North Carolina State University und der Biotechforma Benson Hill, hat Beisels Forschungsgruppe PAMs charakterisiert, die durch eine Reihe von technologisch wichtigen Cas-Nukleasen namens Cas12a erkannt werden. Sie fanden heraus, dass zwei der charakterisierten Nukleasen ganz unterschiedliche PAMs erkannten, obwohl die Nukleasen nahezu identisch waren. Als die Forscher eine Nuklease so mutierten, dass sie wie die andere aussah, entdeckten sie außerdem eine Variante, die verschiedene PAMs erkannte. Diese Nuklease bearbeitete das Genom auch effizienter als die beiden ursprünglichen Nukleasen.
Diese Technologien bieten auch kostengünstige und schnelle Möglichkeiten in der Diagnostik, etwa für den Nachweis von SARS-CoV-2
In der Annahme, dass sich ähnliche Nukleasen ähnlich verhalten, untersuchen Forscher bei der Suche nach neuen und interessanten Cas-Nukleasen normalerweise Nukleasen, die sich extrem voneinander unterscheiden. „Unsere Arbeit stellt diese Annahme infrage mit der überraschenden Erkenntnis, dass sich ähnliche Nukleasen in der Tat ganz anders verhalten können“, sagt Beisel. Aufbauend auf diesem Befund plädieren die Forscher für einen tiefergehendes „Schürfen“ von Nukleasen in der Natur und für die Mutation bestehender Nukleasen zur Veränderung des PAM. Für die in der aktuellen Studie entdeckte Nuklease-Mutante mit einem einzigartigen PAM sieht Beisel technologische Anwendungsmöglichkeiten. Sie ermöglicht der Technologie den Zugang zu noch mehr potenziellen Zielen im Genom und ist mit ihrer erhöhten Aktivität besonders nützlich.
CRISPR-Technologien werden für die Gentherapie zur Heilung von Erbkrankheiten und für die Immuntherapie zur Krebsbekämpfung eingesetzt. „Diese Technologien bieten auch kostengünstige und schnelle Möglichkeiten in der Diagnostik, etwa für den Nachweis von SARS-CoV-2. Unsere Arbeit trägt dazu bei, die Zahl der potenziellen Zielorte im Genom und damit die Flexibilität bei der Anwendung dieser Technologien zu erweitern“, sagt Beisel. Seine Forschungsgruppe erforscht die natürliche Vielfalt der CRISPR-Nukleasen und des CRISPR-Cas-Immunsystems weiter. Damit wollen die Forscher neue und interessante Funktionen aufdecken, die die nächste Revolution der CRISPR-Technologien herbeiführen könnten.
Originalpublikation:
Thomas Jacobsen, Fani Ttofali, Chunyu Liao, Srinivas Manchalu, Benjamin N Gray, Chase L Beisel: Characterization of Cas12a nucleases reveals diverse PAM profiles between closely-related orthologs. Nucleic Acids Research. 2020; DOI: 10.1093/nar/gkaa272.