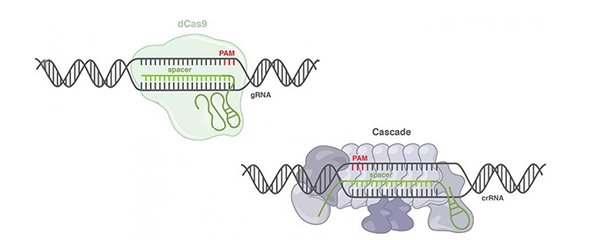
Bakterielle CRISPR-Cas-Systeme können aufgrund der beteiligten Proteine in zwei Klassen unterschieden werden. CRISPR-Systeme der Klasse II benötigen nur ein einzelnes Protein, um die DNA zielgerichtet zu schneiden. Bei der Klasse I müssen hingegen mehrere Proteine in einem sogenannten Cascade-Komplex interagieren. Aufgrund des einfachen Aufbaus wurden Klasse II-Systeme stärker erforscht und sind bisher das Mittel der Wahl für gezielte DNA-Veränderungen. Sie stellen allerdings nur 10% aller in der Natur vorkommenden Systeme dar. „Es gibt also ein großes Repertoire an kaum untersuchten Systemen, von denen wir noch gar nicht wissen, welche Anwendungsmöglichkeiten sie bieten“, sagt Prof. Chase Beisel, Leiter der HIRI-Forschungsgruppe „Biologie synthetischer RNA“. Gemeinsam mit Wissenschaftlern der Duke University und der North Carolina State University hat Beisel erstmals ein CRISPR-Cas-System der Klasse I zum gezielten Verändern der Genexpression menschlicher Zellen programmiert. Die Präzision des neuen Systems war dabei vergleichbar mit der Leistung der bisher eingesetzten Systeme. Die Forscher konnten gezielt DNA-Abschnitte ansteuern und weitere beteiligte Proteine versammeln. Dadurch konnten sie die Expression einzelner Gene erhöhen oder verringern. Solch epigenetische Modifikationen sind ein wichtiger Faktor bei der Genregulation und an der Entstehung verschiedenster Krankheiten beteiligt. Die Studie zeigt, dass sich die komplex aufgebauten Klasse I CRISPR-Cas-Systeme als programmierbare Genwerkzeuge eignen. „Mit den Klasse I CRISPR-Cas-Systemen vergrößern wir die Anzahl der verfügbaren Genwerkzeuge und erschließen möglicherweise ganz neue Anwendungsbereiche“, sagt Beisel.
Eine englischsprachige Meldung der Duke University finden Sie hier.
Originalpublikation:
Adrian Pickar-Oliver, Joshua B. Black, Mae M. Lewis, Kevin J. Mutchnick, Tyler S. Klann, Kylie A. Gilcrest, Madeleine J. Sitton, Christopher E. Nelson, Alejandro Barrera, Luke C. Bartelt, Timothy E. Reddy, Chase L. Beisel, Rodolphe Barrangou & Charles A. Gersbach: Targeted transcriptional modulation with type I CRISPR–Cas systems in human cells. Nat Biotechnol. 2019 DOI: 10.1038/s41587-019-0235-7