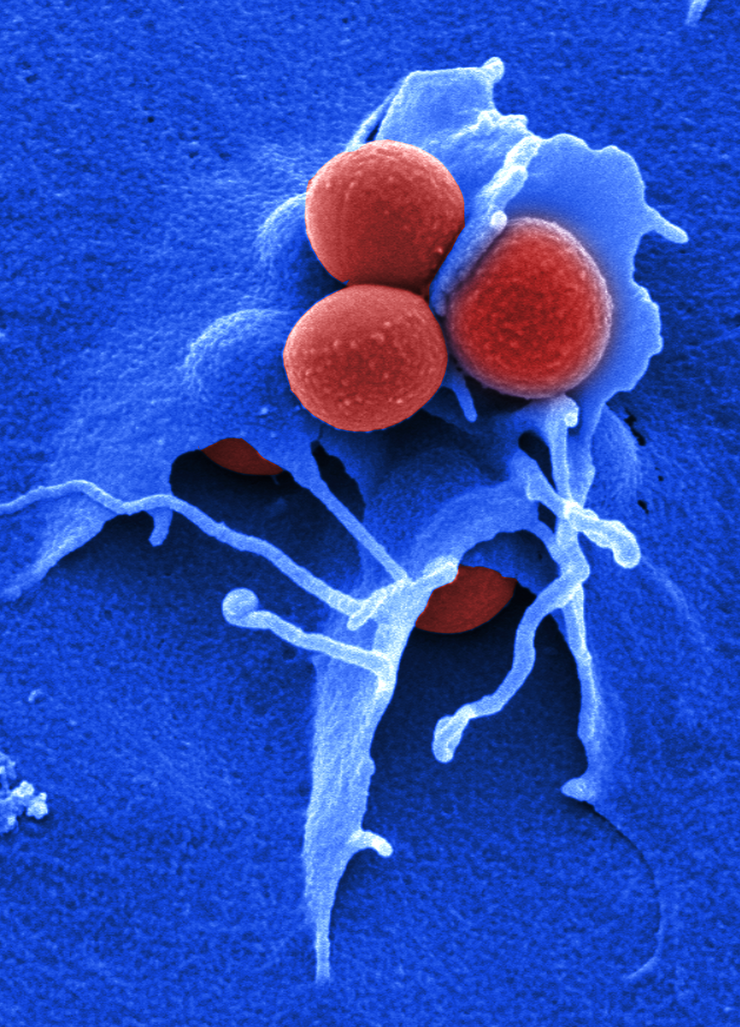
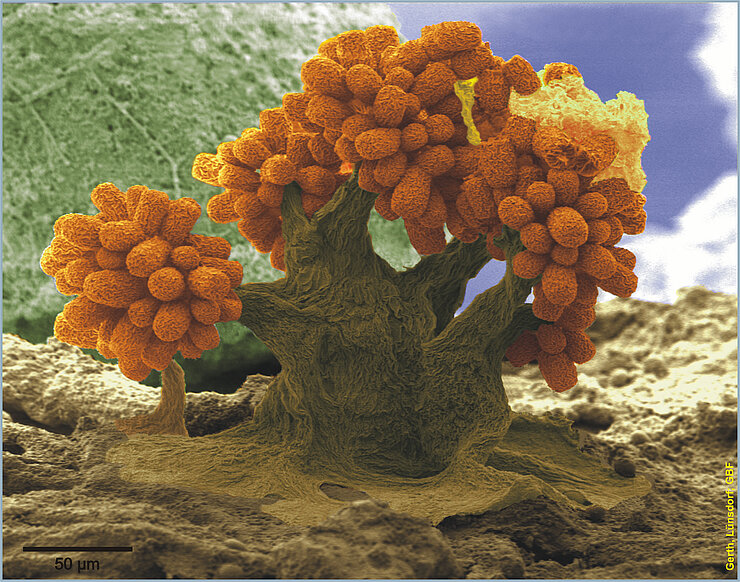
Prof. Rolf Müller, Sprecher des Forschungsbereiches „Neue Wirkstoffe gegen Infektionen“ am Helmholtz-Zentrum für Infektionsforschung (HZI) und geschäftsführender Direktor des Helmholtz-Instituts für Pharmazeutische Forschung Saarland (HIPS) in Saarbrücken, fasst die Lage zusammen: „Antibiotikaresistenzen sind ein globales Problem. Es wird angenommen, dass im Jahr 2050 mehr Todesfälle durch Antibiotika-Resistenzen als durch Krebs verursacht werden könnten. Leider wurde in den letzten zehn Jahren gegen die multiresistenten Erreger kaum etwas unternommen, die derzeit in unseren Krankenhäusern die größten Probleme verursachen. Es ist daher dringend notwendig, dass neue, effektive Medikamente entwickelt werden.“
Im Wettlauf mit den Keimen
Fokus Krankenhaus – Nachholbedarf in der Diagnostik
Die Medizinerin Prof. Susanne Häußler, Leiterin der „Molekularen Bakteriologie“ am HZI, schätzt die Situation ebenfalls sehr kritisch ein: „Die Anzahl der multiresistenten Erreger in den Kliniken nimmt zu, während sich gleichzeitig die neu zugelassenen Medikamente drastisch reduzieren.“ Laut Häußler habe die medizinische Mikrobiologie noch ihre Hausaufgaben zu machen, denn die üblichen Resistenztests von Bakterien in der Klinik beruhen ausschließlich auf dem Wachstum in Anwesenheit von Antibiotika. Das kann manchmal zu lange dauern. Zudem bekommen die Forscher keinerlei Informationen über die Resistenzmechanismen und die klonale Identität der Erreger. „Deshalb arbeiten wir an einer molekularen Diagnostik: Wir isolieren bakterielles Erbmaterial – RNA und DNA – und bestimmen genetische Marker der Resistenz. Außerdem nehmen wir eine genaue Typisierung vor“, sagt Häußler. „Dadurch wird es möglich, schneller und zielgerichteter zu verfolgen, wie sich multiresistente Stämme verbreiten. Mediziner können dann entsprechende prophylaktische und therapeutische Maßnahmen ergreifen.“
Antibiotikaforschung – für Pharmafirmen unattraktiv
Gegen die resistenten Erreger helfen meist nur Antibiotika, die noch nicht in Massen in Gebrauch sind. Doch diese neuen Medikamente sind entweder sehr teuer – oder sie existieren noch gar nicht. Seit 1987 wurde lediglich eine neue Wirkstoffklasse gegen gramnegative Erreger entwickelt – mit bisher nur einem einzigen Wirkstoff. Dazu kommt, dass Pharmaunternehmen kaum noch in die Entwicklung neuer Antibiotika investieren, weil diese Produkte nur selten und über einen kurzen Zeitraum zum Einsatz kommen und somit keinen Profit bringen. Zuletzt hat sich der Pharmakonzern Novartis im Juli 2018 aus der Antibiotika- und Infektionsforschung zurückgezogen.
„Ohne einen Wandel in der medizinischen Forschung und Entwicklung könnte es für Krankheiten, die heute behandelbar sind, in einigen Jahren keine Heilung mehr geben“, sagt Rolf Müller. „In die Forschungslücke müssen nun außeruniversitäre Forschungseinrichtungen wie das HZI und andere Institutionen springen, die vielversprechende Wirkstoffkandidaten für die kommerzielle Weiterentwicklung durch Pharmafirmen suchen und die chemischen Verbindungen soweit wie möglich optimieren.“
Die Jagd nach neuen Wirkstoffen
Die Natur ist die beste Ressource für neue Wirkstoffe. „Etwa 80 Prozent der Antibiotika stammen aus der Natur. Eine schier unerschöpfliche Quelle für neue Wirkstoffe bilden beispielsweise Pilze. Sie produzieren Antibiotika natürlicherweise, um sich gegen Bakterien durchzusetzen und deren Angriffe zu überleben“, sagt Prof. Marc Stadler, Leiter der HZI-Abteilung „Mikrobielle Wirkstoffe“. Auch Myxobakterien, die im Boden leben, sind bekannte, aber noch wenig untersuchte Produzenten von bestimmten Sekundärstoffen. Mittlerweile verfügen die Sammlungen des HZI und HIPS über mehr als 11.000 Stämme von Myxobakterien. Auf der Suche nach neuen Wirkstoffen analysieren die Forscher die Erbinformation der Mikroorganismen und können so abschätzen, ob sie überhaupt das Potenzial zur Wirkstoffbildung haben. Neben Verbindungen aus Pilzen und Myxobakterien enthält die Bibliothek auch zahlreiche Proben aus Actinobakterien, der Organismengruppe, aus der historisch gesehen die meisten Antibiotika stammen. Gelingt es, eine Substanz in größerem Maßstab herzustellen, dann testen die Forscher sie in verschiedenen Assays auf interessante Wirkungen gegen bakterielle und pilzliche Problemkeime und Krebszelllinien. Alle Stoffe landen schließlich in einer Naturstoff-Bibliothek und können so immer wieder auf weitere Wirkungen abgeklopft werden. Müllers Team am HZI und am HIPS entdeckte unter all diesen Substanzen bereits neue Antibiotika-Kandidaten, zum Beispiel die Cystobactamide, die besonders schwer zu bekämpfende gramnegative Erreger abtöten. Ein Wirksamkeitsnachweis bei Infektionen im Tiermodell wurde bereits erbracht. Gemeinsam mit den Braunschweiger HZI-Kollegen und Partnern aus der pharmazeutischen Industrie optimierten die Forscher zudem den Wirkstoff Griselimycin, der gegen den Tuberkulose-Erreger eingesetzt werden könnte, und klärten auch seinen Wirkmechanismus auf. Der Wirkstoff zeigte im Tiermodell hervorragende Aktivität gegen den Erreger der Tuberkulose und ist gegen alle bislang bekannten multiresistenten Keime aktiv.
Allerdings eignen sich die Stoffe, die gut gegen Bakterien wirken, nicht automatisch für die Anwendung am Menschen. Oft ist es ein langer und steiniger Weg in die Medizin – nur wenige Kandidaten sind am Ende erfolgreich. Erschwerend kommt hinzu, dass sie in ausreichenden Mengen in biotechnologischen Verfahren herstellbar sein müssen. In vielen Fällen ist es der Biotechnologieplattform am HZI gelungen, nachhaltige Prozesse zu entwickeln, um die gewünschten Moleküle im Multi-Gramm-Maßstab herzustellen. Aber manchmal ist es nicht kostengünstig möglich. An dieser Stelle kommen die synthetische Biologie und die medizinische Chemie ins Spiel, die die häufig sehr komplexen Strukturen der Naturstoffe durch einfachere und leichter zu optimierende Moleküle ersetzen. Diese lassen sich dann weiter anpassen, um beispielsweise eine bessere Verträglichkeit, eine höhere Stabilität im Blutkreislauf oder höhere Wirkspiegel am Infektionsort zu erzielen.
Erreger entwaffnen statt töten

Neben neuen Antibiotika gibt es auch alternative Methoden, um den resistenten Erregern zu Leibe zu rücken. Seit einigen Jahren weckt die Antivirulenz-Strategie große Hoffnungen: Ihr Ziel ist es, die Bakterien zu entwaffnen, statt sie zu töten. Dr. Eva Medina, Leiterin der HZI-Arbeitsgruppe „Infektionsimmunologie“, sucht speziell nach Wirkstoffen, die verhindern, dass die Bakterien krankmachende Proteine – sogenannte Virulenzfaktoren – bilden. Dabei handelt es sich zum Beispiel um Proteine, mit deren Hilfe Erreger in Wirtszellen eindringen oder sich gegen das Immunsystem wehren können. „Der große Vorteil dieser Strategie ist es, dass die Bakterien am Leben bleiben und weiter vermehrungsfähig sind. Dadurch haben diejenigen unter ihnen, die eine Resistenz gegen einen Antivirulenz-Wirkstoff entwickeln, keinen besonderen Vorteil – und können sich daher auch nicht stärker ausbreiten als ihre Artgenossen.“ Die Entwicklung von Antivirulenzmitteln ist damit auch gewinnträchtiger für die Pharmaindustrie, weil diese Medikamente eben nicht ihre Wirkung durch entstehende Resistenzen einbüßen und so länger einsetzbar bleiben. Allerdings wirken die Antivirulenz-Medikamente immer nur sehr spezifisch bei einem Erreger. Das setzt eine sehr genaue Diagnostik auch bei lebensgefährlich erkrankten Patienten voraus, die einer gewissen Zeit bedarf. Im Mausmodell hat das Team um Eva Medina zudem herausgefunden, dass die genetische Ausstattung und das Immunsystem einen Einfluss darauf haben, wie krankheitserregend ein Keim für den einzelnen Patienten wirklich ist. „Wahrscheinlich muss man die Antivirulenz-Strategie auch individuell an die Patienten anpassen“, sagt Medina.
Innovativer Ansatz – programmierbare RNA-Antibiotika
Die heutigen Antibiotika wirken meist in der Breite: Sie attackieren entweder gramnegative oder grampositive Bakterien und zerstören beiläufig viele harmlose oder nützliche Mikroorganismen gleich mit. Für bestimmte Infektionskrankheiten kann es jedoch notwendig sein, im Mikrobiom des Menschen – zum Beispiel in der Darmflora – eine ganz bestimmte Bakterienart auszuschalten. Prof. Jörg Vogel, Gründungsdirektor des Helmholtz-Instituts für RNA-basierte Infektionsforschung (HIRI) in Würzburg, verfolgt dazu einen neuen Forschungsansatz auf Basis von RNA-Molekülen – das sind Abschriften der Erbinformation. RNA-Moleküle bestimmen alle möglichen Prozesse in der Zelle und stehen auch im Zusammenhang mit vielen Krankheiten. Vogels Ziel: Im Sinne des Patienten sollen der Erreger zurückgedrängt und der Wirt besser gegen die Infektion geschützt werden. In beiden Fällen spielen RNA-Moleküle eine wichtige Rolle, etwa wenn Erreger ihre Umgebung abtasten, um den richtigen Moment für den Wirtsbefall zu entscheiden. „Mit RNA-Molekülen können wir programmierbare Medikamente entwickeln und damit im Gegensatz zu Antibiotika auch gezielt eine bestimmte Bakterienart angreifen. Andere, für den Menschen nützliche Arten bleiben so verschont“, sagt Vogel. „Mit einer von uns entwickelten Methode können wir im Detail beobachten, welche Gene eines Erregers bei einer Infektion an- oder abgeschaltet sind und was daraufhin im Wirt passiert. Diese Erkenntnisse liefern uns völlig neue Angriffspunkte für Therapien.“
Besonders im Visier haben die RNA-Forscher derzeit den Erreger Fusobacterium nucleatum, der normalerweise in der Mundhöhle vorkommt, aber auch mit dem Speichel beim Schlucken in den Darm gelangt. Seit etwa sieben Jahren ist bekannt, dass die Fusobakterien sich an Darmkrebsvorläuferzellen anheften. „Die Bakterien scheinen zwar nicht ursächlich an der Darmkrebsbildung beteiligt zu sein, sie mindern aber dramatisch die Behandlungschancen, indem sie die Chemotherapeutika inaktivieren“, sagt Vogel. „Mit einem programmierbaren Antibiotikum könnten wir ganz gezielt nur die Fusobakterien ausschalten.“ Momentan gibt es bei den programmierbaren RNA-Antibiotika noch viele offene Fragen, aber im Prinzip ist deren Wirkungsweise bereits bestätigt, und Vogel sieht in ihnen ein großes Potenzial auch für die Korrektur von nach Krankheit veränderter Darmflora.
Autorin: Susanne Thiele
Veröffentlichung: November 2018